

Цель работы
Изучить способы визуализации и наглядного представления данных из выборки в R.
Выполнение работы
1) Для построения первого графика использовали встроенный набор данных, доступный по команде data(InsectSparays). Построили график с boxplot’а ми по этим данным. Указали заголовки осей и самого рисунка:
•Аргумент xlab – “Инсектициды”
•Аргумент ylab – “Количество выживших насекомых”
•Аргумент main – “Эффективность инсектицидов”
Также каждый отдельно взятый boxplot на графике был закрашен своим цветом. Рассчитали среднее значение для каждого boxplot’а и добавить эти средние значения в c соответствующий boxplot с помощью функции точек points().
Код в соответствии с листингом 1 и результат в соответствии с рисунком 1.
Листинг 1 – Код пункта 1
# Пункт 1
library(datasets)
data(InsectSprays)
force(InsectSprays)
#Отображенеи графика boxplot(count ~ spray,
data = InsectSprays, xlab = "Инсектициды",
ylab = "Количество выживших насекомых", main = "Эффективность инсектицидов",
col = c("red", "green", "blue", "brown", "blueviolet", "cyan"))
#Определение средненго значеиня каждого boxplot
mean_point <- lapply(unstack(InsectSprays), mean)
# Отбражение средненго значеиня boxplot на графике points(1:length(mean_point), mean_point, col = "black", pch = 20)
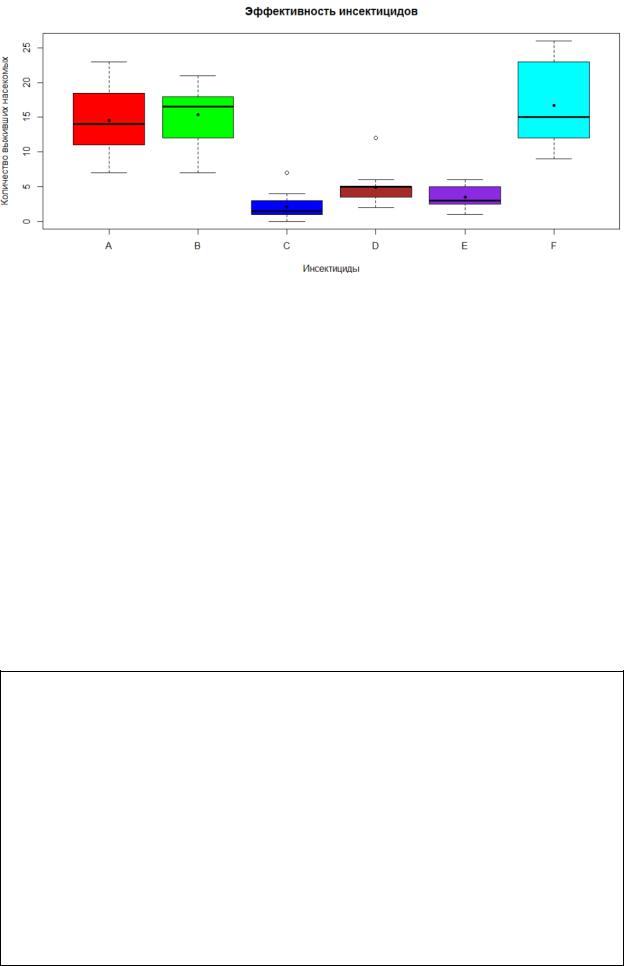
Рисунок 1 – График пункта 1
2) Для построения следующего графика использовали встроенный набор данных, доступный по команде data(ToothGrowth) и пакет “ggplot2”. Для начала подготовили данные для отображения violin plot. Переменную “dose”
перевели в переменную с типом factor. Затем отобразили violin plot, так чтобы по оси X была отложена переменная “dose”, а по оси Y – “len”. Внутри каждого violin plot’а добавили среднее значение и стандартное отклонение. Линии окантовки графиков сделали разных цветов. Изменили отображение позиции легенды на отображение под графиком.
Код в соответствии с листингом 2 и результат в соответствии с рисунком 2.
Листинг 2 – Код пункта 2
#Пункт 2
#Подключение необходимых пакетов library(datasets)
library(ggplot2)
#Получение датасета data(ToothGrowth) force(ToothGrowth)
#Приведене ToothGrowth$dose к фактору
ToothGrowth$dose=as.factor(ToothGrowth$dose)
#Вычисляем среднее значение и стандартное отклонене mean_tg <- aggregate(len ~ dose, ToothGrowth, mean) sd_tg <- aggregate(len ~ dose, ToothGrowth, sd)
2

# Отображение графика
ggplot(ToothGrowth, aes(x=dose, y =len, col=dose)) + geom_violin(trim = FALSE) +
theme_light()+
geom_point(data = sd_tg, aes(dose, mean_tg$len-len))+ geom_point(data = sd_tg, aes(dose, mean_tg$len+len))+ geom_point(data = sd_tg, aes(dose, mean_tg$len), cex=4)+ theme(legend.position="bottom")
Рисунок 2 – График пункта 2
3)Следующие графиками гистограммы распределений. Возьмем 2
разных закона распределения вероятностей. Отобразим на графике гистограмму распределения точек (моделируем по 500 значений). Столбцы гистограммы сделали разными цветами отличными от стандартных. Над каждым столбцом гистограммы отобразили количество находящихся в ней точек. Для этого используем функцию text(). На гистограмму наложили кривую используемого распределения. Также дали наименование осям и самому графику.
Код в соответствии с листингом 3 и результат в соответствии с рисунками 3-4.
Листинг 3 – Код пункта 3
# Пункт 4
num_count = 500 # Кол-во значений выборки
# Формирование выборки экспоненциального и нормального распределений exp_sample = rexp(num_count)
norm_sample = rnorm(num_count)
3

#Рисуем график эксп. распред. hist(exp_sample,
labels = TRUE, # Отображает кол-во точек в ситолбце main="Экспоненциальное распределение",
xlab="x", ylab="Количество", col="cyan")
#Рисуем кривую эксп. распред.
curve(dexp(x)*num_count/2, add = T,
col = "blueviolet", lwd = 2)
#Рисуем на графике график норм. распред. hist(norm_sample,
labels = TRUE, # Отображает кол-во точек в ситолбце main="Нормальное распределение",
xlab="x", ylab="Количество", col="cyan")
#Рисуем на графике кривую норм. распред. curve(dnorm(x)*num_count/2,
add = T,
col = "blueviolet", lwd = 2)
Рисунок 3 – График экспоненциального распределения пункта 3
4

Рисунок 4 – График нормального распределения пункта 3
5

4) Следующим отобразили график 3D Pie chart. Воспользуемся пакетом “plotrix”. Для построения используем набор данных iris. Каждая область графика должна иметь свой цвет, название и находиться на некотором расстоянии от соседних областей. Также необходимо отобразить название самого графика.
Код в соответствии с листингом 4 и результат в соответствии с рисунком 5.
Листинг 4 – Код пункта 4
#Пункт 4
#Подключение необходимых пакетов library(plotrix)
#Создаем данне для построения pie графика
temp <- aggregate(Sepal.Length ~ Species, iris, mean)
# Рисуем pie график
mtcars_plot <- pie3D(temp$Sepal.Length, radius = 1,
labels = temp$Species, explode = 0.1, col=rainbow(3),
main = "3D Pie chart of iris")
Рисунок 5 – График пункта 4
Код программы целиком представлен в Приложении А.
6
Вывод
Выполнив в данную лабораторную работу, мы изучили и получили практические навыки в работе с визуализации и наглядного представления данных из выборки в R. Для этого мы воспользовались пакетами datasets
(предоставляет набор датасетов), ggplot2 (пакет визуализации данных) и plotrix (пакет визуализации данных). С их помощью нами были построены 5
графиков визуализирующих данные из датасетов в соответствии с поставленным заданием.
7
Приложение А. Листинг программы
#лаба 4
#Пункт 1
library(datasets)
data(InsectSprays)
force(InsectSprays)
#Отображенеи графика boxplot(count ~ spray,
data = InsectSprays, xlab = "Инсектициды",
ylab = "Количество выживших насекомых", main = "Эффективность инсектицидов",
col = c("red", "green", "blue", "brown", "blueviolet", "cyan"))
#Определение средненго значеиня каждого boxplot
mean_point <- lapply(unstack(InsectSprays), mean)
# Отбражение средненго значеиня boxplot на графике points(1:length(mean_point), mean_point, col = "black", pch = 20)
########################################
#Пункт 2
#Подключение необходимых пакетов library(datasets)
library(ggplot2)
#Получение датасета data(ToothGrowth) force(ToothGrowth)
#Приведене ToothGrowth$dose к фактору
ToothGrowth$dose=as.factor(ToothGrowth$dose)
#Вычисляем среднее значение и стандартное отклонене mean_tg <- aggregate(len ~ dose, ToothGrowth, mean) sd_tg <- aggregate(len ~ dose, ToothGrowth, sd)
#Отображение графика
ggplot(ToothGrowth, aes(x=dose, y =len, col=dose)) +
8
geom_violin(trim = FALSE) + theme_light()+
geom_point(data = sd_tg, aes(dose, mean_tg$len-len))+ geom_point(data = sd_tg, aes(dose, mean_tg$len+len))+ geom_point(data = sd_tg, aes(dose, mean_tg$len), cex=4)+ theme(legend.position="bottom")
########################################
# Пункт 4
num_count = 500 # Кол-во значений выборки
#Формирование выборки экспоненциального и нормального распределений exp_sample = rexp(num_count)
norm_sample = rnorm(num_count)
#Рисуем график эксп. распред.
hist(exp_sample,
labels = TRUE, # Отображает кол-во точек в ситолбце main="Экспоненциальное распределение",
xlab="x", ylab="Количество", col="cyan")
#Рисуем кривую эксп. распред. curve(dexp(x)*num_count/2,
add = T,
col = "blueviolet", lwd = 2)
#Рисуем на графике график норм. распред. hist(norm_sample,
labels = TRUE, # Отображает кол-во точек в ситолбце main="Нормальное распределение",
xlab="x", ylab="Количество", col="cyan")
#Рисуем на графике кривую норм. распред. curve(dnorm(x)*num_count/2,
add = T,
col = "blueviolet", lwd = 2)
9
########################################
#Пункт 4
#Подключение необходимых пакетов library(plotrix)
#Создаем данне для построения pie графика
temp <- aggregate(Sepal.Length ~ Species, iris, mean)
# Рисуем pie график
mtcars_plot <- pie3D(temp$Sepal.Length, radius = 1,
labels = temp$Species, explode = 0.1, col=rainbow(3),
main = "3D Pie chart of iris")
10
